Talaan ng mga Nilalaman:
- Hakbang 1: Simulan ang Graphical User Interface (GUI)
- Hakbang 2: Naglo-load at Nagbabasa ng Mga Imahe ng MRI sa MATLAB
- Hakbang 3: Pagsala ng Larawan
- Hakbang 4: Paghiwalay ng Tumor Sa Pamamagitan ng Elliptical Mask
- Hakbang 5: Paglabas ng Tumor
- Hakbang 6: Sinusuri ang Mga Physical Properties ng Mga Tumors
- May -akda John Day day@howwhatproduce.com.
- Public 2024-01-30 13:13.
- Huling binago 2025-01-23 15:13.
Ni: Madhumita Kannan, Henry Nguyen, Ashley Urrutia Avila, Mei Jin
Ang MATLAB code na ito ay isang programa upang makita ang eksaktong sukat, hugis, at lokasyon ng isang tumor na matatagpuan sa pag-scan sa utak ng pasyente ng MRI. Ang program na ito ay idinisenyo upang orihinal na gumana sa pagtuklas ng tumor sa pag-scan sa utak ng MRI, ngunit maaari rin itong magamit para sa mga diagnostic ng kanser sa iba pang mga pag-scan ng organ din.
Ang mga sumusunod na tagubilin ay unang ilalarawan ang mga pamamaraan para sa pagtatasa ng imahe sa pamamagitan ng pag-filter at paglilinis ng MRI scan, sa pamamagitan ng binarizing, median filtering, at sliding windows. Susunod, magtuturo ito sa kung paano ihiwalay ang tumor gamit ang isang paunang nabuo na elliptical mask, at sinala ito ng karagdagang upang ibalangkas ang perimeter ng hugis ng tumor.
Kapag nakita ang tumor, ang mga tagubilin ay higit na ilalarawan kung paano isasama ang program na ito sa isang graphic na interface ng gumagamit (GUI). Sa buong mga tagubiling ito, ang naaangkop na code at mga file ay ikakabit upang makatulong na ipaliwanag kung paano gumagana ang pagsusuri sa pag-scan ng MRI na ito.
Ang ilang mga bagay na dapat malaman, i-download, at maghanda bago ka magpatuloy sa itinuturo na ito: 1. Tiyaking na-download ang pinakabagong bersyon ng MATLAB. Maaari mong i-install ang R2018b dito:
2. Upang patakbuhin ang program na ito, kailangan mong magkaroon ng pag-access sa mga file ng pag-scan ng utak ng MRI. Bagaman ang ilan ay laging matatagpuan mula sa mga imahe ng Google, ang masusing at tumpak na pagsusuri ay maaaring gampanan mula sa tamang mga imahe ng iba't ibang mga layer ng pag-scan sa utak para sa bawat pasyente. Maaari mong ma-access ang mga file para sa 20 magkakaibang mga pasyente na may glioblastoma pre at i-post ang paggamot mula sa database na ito:
3. Ang pokus ng program na ito at ang iba't ibang mga pamamaraan na gumagabay sa proyektong ito ay nakabalangkas sa papel ng pagsasaliksik na ito:
Hakbang 1: Simulan ang Graphical User Interface (GUI)
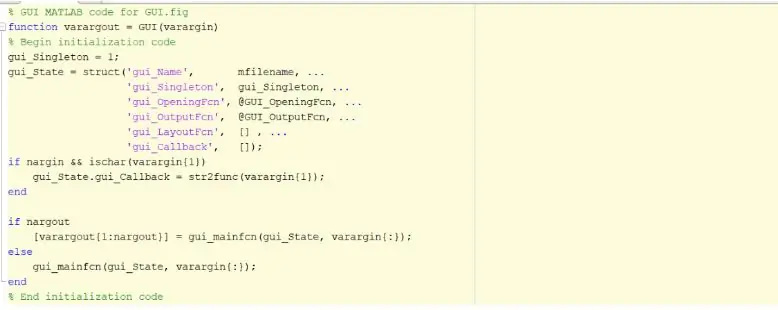
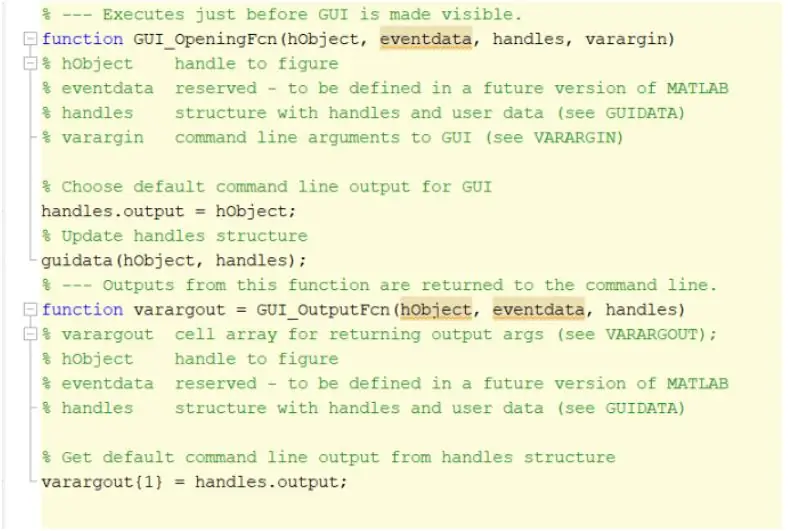
Ang unang hakbang ay upang lumikha at simulan ang graphic na interface ng gumagamit, GUI. Magagawa ito sa pamamagitan ng gabay sa pagta-type sa window ng utos, pagpindot sa enter, at paglikha ng isang bagong GUI. Kapag nakumpleto ang hakbang na ito maaari kang magsimulang lumikha ng mga pag-andar tulad ng mga palakol, static na teksto, i-edit ang teksto, at mga pindutan ng itulak na ipapakita sa sandaling ang programa ay tumakbo at ang gumagamit ay maaaring makipag-ugnay. Ang mga pagpapaandar na ito ay maaaring mai-edit at manipulahin sa pamamagitan ng inspektor ng pag-aari, subalit ang pinakamahalagang tampok na dapat mabago kapag lumilikha ng mga pagpapaandar na ito ay ang pangalan ng Tag. Mahalagang palitan ang pangalan ng Tag ng bawat pagpapaandar na ipinatupad sapagkat papayagan kaming lumikha ng isang kilalang function ng callback. Kapag nasiyahan ka sa layout ng iyong GUI maaari kang magpatuloy sa paglo-load ng mga DICOM file na ipapakita sa loob ng GUI.
Hakbang 2: Naglo-load at Nagbabasa ng Mga Imahe ng MRI sa MATLAB
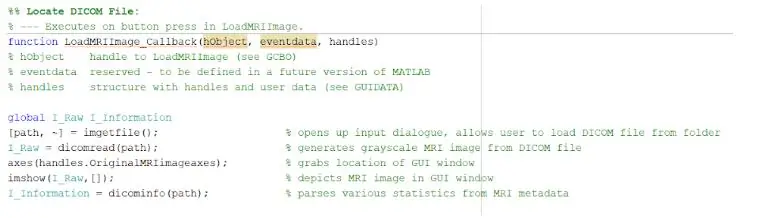
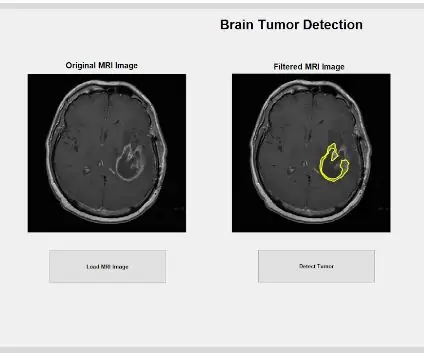
Upang mai-load ang mga file ng DICOM kakailanganin mong wastong ipasimuno ang function na call back na naisakatuparan kapag pinindot ang pindutan na "Load MRI Image." Kapag nakumpleto na ito, dapat kang lumikha ng isang pandaigdigang variable na magpapakita ng imahe sa mga humahawak na palakol kung saan nais mong ipakita ang Orihinal na Imahe ng MRI. Ang mga imahe ng pag-scan ng MRI na na-download mula sa database ay lahat ng mga naka-format na file ng DICOM na kailangang mai-load sa iyong direktoryo ng MATLAB. Hanapin ang file gamit ang imgetfile upang mai-load ang mga ito sa programa. Nabasa ang mga imahe gamit ang built in MATLAB function na 'dicomread', at ang unang hilaw na imahe para sa bawat file ay naka-embed sa kaliwang GUI axe gamit ang imshow.
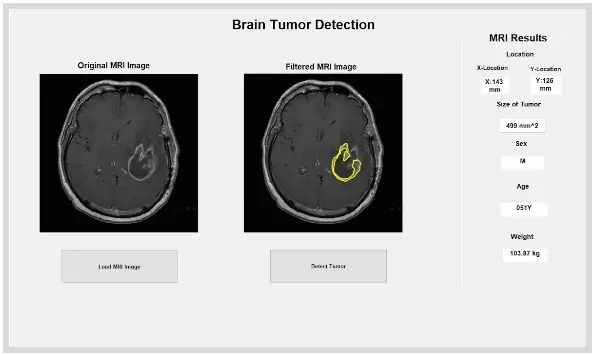
Ang built in na MATLAB function na ‘dicominfo’ ay lubhang kapaki-pakinabang din sa pagtugon sa lahat ng impormasyon ng bawat MRI dicom file. Ginamit namin ang pagpapaandar na ito upang makuha ang lahat ng naglalarawang impormasyon ng mga pasyente, tulad ng kanilang kasarian, edad, timbang, at taas. Ang pagpapaandar na ito ay nagbibigay sa iyo ng order ng stack na kung saan ay kapaki-pakinabang para sa pagpapatupad ng programa sa loob ng graphic na interface ng gumagamit. Lumikha kami ng mga variable para sa bawat isa sa naglalarawang impormasyon ng mga pasyente na gagamitin para sa GUI kapag pinindot ang pindutang makita.
Hakbang 3: Pagsala ng Larawan

Kapag ang DICOM file ng hilaw na imahe ay na-load at nabasa na, ang imahe ay kailangang mai-convert mula sa grayscale sa isang binarized form na binubuo lamang ng mga itim at puting mga pixel Ginamit namin ang function na 'imbinarize' upang lumikha ng isang binary na imahe mula sa hilaw na imahe ng pagkontrol sa mga aspeto ng adaptive thresholding sa sensitibong halaga na 0.59. Ang default na kadahilanan ng pagiging sensitibo sa threshold, ang 0.5 ay mababa at hindi makita ang mas maliwanag na mga bloke at mga spot mula sa imahe, kaya nadagdagan namin ito sa 0.59.
Pagkatapos ay naproseso ang binarized na imahe sa pamamagitan ng isang median filter gamit ang pagpapaandar na 'medfilt2' dahil ang binarized na imahe ay dalawang-dimensional. Itinakda namin ang bawat output pixel upang maglaman ng median na halaga sa 5 x 5 na kapitbahayan sa paligid ng kaukulang pixel sa input na binarized na imahe. Binabawasan nito ang ingay at pinapanatili ang mga gilid sa isang 5 x 5 parisukat sa paligid ng bawat pixel. Susunod, inilalapat namin ang isang sliding window gamit ang 'strel', upang lumikha ng isang hugis ng disk na flat na elemento ng pagbubuo na may radius ng kapitbahayan na 2 upang makilala ang bawat gitnang, pinagmulan ng pixel, sa bawat kapitbahayan ng disk. Gumamit kami ng isang elemento ng pagbubuo ng disk dahil sinusuri namin ang bawat pabilog na lugar at ang mga pixel sa loob ng bawat lugar, kaya't ang isang elemento ng hugis ng disk ay mas kapaki-pakinabang.
Kapag na-filter ang imahe, maaari itong malinis gamit ang function na 'imclose' upang alisin ang mga itim na spot sa pagitan ng mga na-filter na puting pixel sa imahe, at isara ang lahat ng mga puwang sa paligid nito. Ang kumpletong naprosesong imahe ay maaaring mai-plot sa pangalawang subplot ng paunang inilalaan na pigura, na pinapayagan ang isang paghahambing sa pagitan ng hilaw at na-filter na imahe.
Hakbang 4: Paghiwalay ng Tumor Sa Pamamagitan ng Elliptical Mask


Ang mga maliwanag na spot ng bukol ay maaaring ihiwalay mula sa pangunahing nasala na imahe sa pamamagitan ng isang paunang nabuo na elliptical mask. Upang likhain ang maskara na ito, dapat mong malaman ang laki ng orihinal, hilaw na imahe ng pag-scan ng MRI, at ang paggamit nito sa haba ng hilera at haligi, bilang x at y-coordinate ayon sa pagkakabanggit, naglalaan ng mga coordinate para sa elliptical. Itinakda namin ang y-axis bilang isang pangunahing axis na may radius na 50 mga yunit mula sa gitna, at ang menor de edad na axis na may isang radius na 40 mga yunit mula sa gitna.
Ginamit namin ang MATLAB function na 'meshgrid' upang makabuo ng isang eroplano na kartesian na may dalawang dimensional na mga coordinate ng grid batay sa mga coordinate na nilalaman sa mga vector mula 1 hanggang sa haba ng x-axis, at mula 1 hanggang sa haba ng y-axis ng imahe. Ang Col ay isang matrix kung saan ang bawat hilera ay isang kopya ng x-axis, at ang Row ay isang matrix kung saan ang bawat haligi ay isang kopya ng y-axis. Ang grid ng cartesian na kinakatawan ng mga coordinate na Col at Row ay may haba (1: Y_Size) na mga hanay at haba (1: X_Size) na mga haligi. Gamitin ang mga indeks ng Col at Row na nabuo ng cartesian grid upang matukoy ang equation ng ellipse depende sa paunang natukoy na radius at center coordinate. Ang elliptical outline ay maaari nang mapunan ng mga puting pixel na matatagpuan mula sa mga spot ng tumor.
Ang paggamit ng paunang nabuo na elliptical mask maaari naming mai-crop ang tukoy na tumor na nais mong pag-aralan mula sa na-filter na imahe. Nakita ng elliptical mask kung aling mga spot ang lohikal na umaangkop sa loob ng balangkas ng ellipse at tinatanggap ito bilang isang spot sa nai-filter na imahe upang tanggapin bilang isang bukol. Ang function na 'bwareafilt' pagkatapos ay sinasala ang lahat ng iba pang mga bagay sa labas ng napansin na tumor na ito mula sa imahe. Gumamit kami ng isang tukoy na window ng 500 ng 4000 empirically batay sa mga sukat ng lahat ng mga imahe. Pagkatapos ay naglapat kami ng isa pang sliding window na may 'strel' bilang isang flat disk na hugis na elemento ng pagbubuo ng isang mas malaking radius ng kapitbahayan na 6, upang isara ang mga puwang sa pagitan ng bawat gitnang puting pixel sa loob ng napansin na bukol. Ang natukoy na spot ng tumor ay karagdagang nalinis gamit ang 'imclose' upang higit na matanggal ang mga itim na pixel at punan ang lahat ng mga butas gamit ang 'imfill'. Ang naprosesong tumor na ito ay maaaring ipakita sa pangatlong subplot sa preallocated plot upang magbigay ng paghahambing sa pagitan ng nakahiwalay na tumor at ng orihinal at nai-filter na mga imahe ng MRI scan.
Hakbang 5: Paglabas ng Tumor
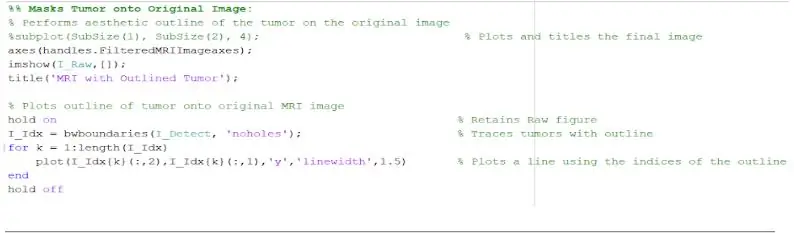
Ngayon na ang bukol ay nakahiwalay sa maskara, maaari itong ibalangkas at maipakita sa orihinal na imahe, upang maipakita ang eksaktong lokasyon nito. Upang magawa ito, ginamit namin ang pagpapaandar na 'bwboundaries' upang subaybayan ang dating napansin na tumor na may isang balangkas. Natukoy namin ang balangkas upang hindi isama ang mga butas sa loob ng bagay ng tumor dahil ito ay binabalangkas. Maaari itong i-plot sa orihinal, hilaw na imahe, gamit ang isang 'for' loop na naglalagay ng balangkas sa paligid ng tumor gamit ang mga indeks ng linya na may isang linya na lapad ng 1.5 pixel. Ang balangkas na ito ay naka-plot sa hilaw na imahe, ipinapakita ang eksaktong sukat at lokasyon ng tumor, na may kaugnayan sa orihinal na pag-scan ng MRI.
Hakbang 6: Sinusuri ang Mga Physical Properties ng Mga Tumors
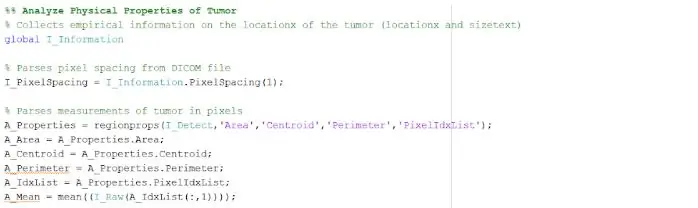
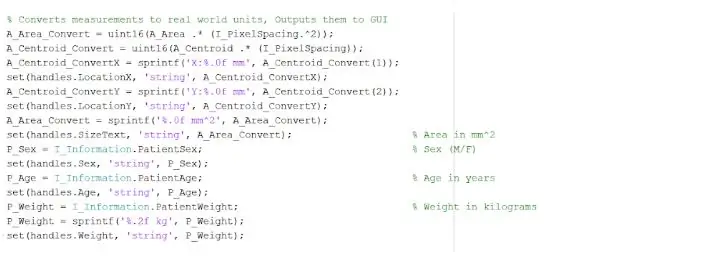
Ang nakahiwalay at nakabalangkas na lugar ay maaaring magbigay sa amin ng kapaki-pakinabang na impormasyon tungkol sa laki, lugar, at lokasyon ng bukol. Ginamit namin ang pagpapaandar ng 'regionprops' upang makita ang mga katangian ng tumor na nauugnay sa lugar, perimeter, centroids, at ang halaga ng pixel index. Binibigyan kami ng halagang pixel index ng mga tunay na yunit ng mundo para sa bawat pixel ng bawat imahe, natatangi sa bawat pag-scan. Ang mga pag-aari na ito ay maaaring mai-convert sa mga yunit ng millimeter ng totoong mundo. Ang impormasyong empirikal na ibinibigay sa amin ng programa ay kakaiba para sa bawat pag-scan ng MRI at lubos na kapaki-pakinabang sa pagtukoy ng laki, lokasyon, at uri ng tumor, na maaaring pag-aralan at isama ng mga gumagamit sa graphic na interface ng gumagamit.
Inirerekumendang:
Pagtuklas sa Mukha sa Raspberry Pi 4B sa 3 Mga Hakbang: 3 Mga Hakbang

Pagtuklas ng Mukha sa Raspberry Pi 4B sa 3 Mga Hakbang: Sa Instructable na ito ay isasagawa namin ang pagtuklas ng mukha sa Raspberry Pi 4 kasama ang Shunya O / S gamit ang Shunyaface Library. Ang Shunyaface ay isang library ng pagkilala / pagkakita sa mukha. Nilalayon ng proyekto na makamit ang pinakamabilis na pagtuklas at bilis ng pagkilala sa
MATLAB Madaling Pagtuklas ng Mukha: 4 na Hakbang

MATLAB Madali na Pagtuklas ng Mukha: Ang pangunahing layunin ng mga itinuturo na ito ay upang maipakita kung gaano kadali, magiging ang pagpoproseso ng imahe, Sa tulong ng pagtuklas ng MATLABFace at pagsubaybay ay isang mahalagang at aktibong larangan ng pagsasaliksik, kaya't bakit ko ipaliwanag kung paano ito magagawa wit
Pagtuklas ng Mga Sitwasyon ng Mga Sumibol - Qualcomm Dragonboard 410c: 7 Mga Hakbang

Pagtuklas ng Mga Sitwasyon ng Mga Emergency - Qualcomm Dragonboard 410c: Naghahanap ng mga sistemang panseguridad na gumagana sa pagsubaybay ng mga sitwasyon ng mga emergency, posible na mapansin na napakahirap iproseso ang lahat ng naitala na impormasyon. Sa pag-iisip tungkol doon, nagpasya kaming gamitin ang aming kaalaman sa pagproseso ng audio / imahe, mga sensor ng
3 Mga Kamangha-manghang Mga Utak ng Control ng BRAIN / MIND Control LedStrip LED Sa Arduino at Neurosky: 6 na Hakbang (na may Mga Larawan)

3 Kamangha-manghang Mga Utak ng Control ng BRAIN / MIND Control LedStrip LED Sa Arduino at Neurosky: Nais mo bang buksan o i-OFF ang mga ilaw sa pamamagitan lamang ng pag-iisip tungkol dito? O nais mong malaman kung gaano ka-stress sa pamamagitan ng pagtingin sa kulay ng isang humantong sa RGB? Habang ngayon maaari mo sa pamamagitan ng pagsunod sa mga Instructionable na ito! Upang makakuha ng isang pakiramdam para sa kung ano ang pupuntahan namin
Mga Paraan ng Pagtuklas sa Antas ng Tubig Arduino Paggamit ng Ultrasonic Sensor at Funduino Water Sensor: 4 na Hakbang

Mga Paraan ng Pagtuklas sa Antas ng Tubig Arduino Gamit ang Ultrasonic Sensor at Funduino Water Sensor: Sa proyektong ito, ipapakita ko sa iyo kung paano lumikha ng isang murang detektor ng tubig gamit ang dalawang pamamaraan: 1. Ultrasonic sensor (HC-SR04) .2. Funduino water sensor
